- RNA-Primer
-
Als Primer (Pl.: die Primer, IPA: [ˊpʁaɪ̯mɐ]) wird in der Molekularbiologie ein Oligonukleotid bezeichnet, das als Startpunkt für DNA-replizierende Enzyme wie die DNA-Polymerase dient.
DNA-Polymerasen benötigen eine Hydroxylgruppe als Startpunkt für ihre erste Verknüpfungsreaktion. Primer stellen mit ihrem 3’OH-Ende eine passende Hydroxyfunktion zur Verfügung. Primer können sowohl aus DNA als auch aus RNA bestehen. Bei der Replikation dient in Prokaryoten und Eukaryoten RNA als Primermaterial. In Prokaryoten synthetisiert die Primase, auch als DnaG-Protein bezeichnet, die Primersequenzen. In Eukaryoten besitzt die DNA-Polymerase α eine Primasefunktion. Einen Sonderfall stellt die DNA-Synthese an den Telomeren eukaryotischer Zellen dar. Hier dient dem polymerisierendem Enzym Telomerase das 3’OH-Ende der DNA als Primersequenz. Bei Prokaryoten werden die bei der Replikation auftauchenden Primer durch 3’-5’Exonukleaseaktivität der Polymerase I oder durch die RNase H entfernt. In eukaryotischen Zellen werden die Primer durch verdrängende DNA-Synthese der die Polymerase δ und Restriktion durch die Flap-Endonuklease entfernt.
Auch bei der In-vitro-Amplifikation von DNA, beispielsweise bei der Polymerase-Kettenreaktion (PCR), der DNA-Sequenzierung oder bei der reversen Transkription, werden Primer benötigt. Hier lässt sich mit Hilfe der Primer der spezifische DNA-Abschnitt, der amplifiziert werden soll, festlegen.
Für die PCR werden deshalb Nukleotidsequenzen, die den zu amplifizierenden DNA-Abschnitt flankieren, bestimmt. Gemäß diesen Sequenzen werden nun passende Primersequenzen synthetisch hergestellt. Ein Primer repräsentiert hierbei jeweils den gegenläufigen Strang zu seinem „Primerpartner“. Primer für PCR-Ansätze haben in der Regel eine Länge von 18-30 Nukleotiden. Diverse Biotechnologiefirmen bieten mittlerweile maßgeschneiderte Primer für molekularbiologische Anwendungen an. Durch maßgeschneiderte Missmatchprimer lassen sich über die PCR-Technik auch gezielt Mutationen in Gene einführen, die z.B. im Austausch einer Aminosäure bestehen.
Primerdesign
Die Wahl einer Sequenz und deren Länge in Basenpaaren für einen Primer hängt von einer Vielzahl an Überlegungen ab. Die Länge einer Primersequenz darf eine bestimmte Länge nicht überschreiten. Mit der Länge des Primers wächst die Schmelztemperatur des Primers linear. Die Schmelztemperatur eines Primers (Tm) ist diejenige Temperatur, bei der der Primer sich von seiner template-DNA löst (schmilzt), oder anders ausgedrückt, unter seiner Schmelztemperatur hybridisiert er mit der template-DNA. Ist die Schmelztemperatur allerdings zu hoch (im allgemeinen über 80°C), können die verwendeten Enzyme wie z.B. die DNA-Polymerase denaturieren. Ist die Temperatur allerdings zu niedrig (unter 55°C) können die Primer unspezifisch an nicht-template-DNA-Sequenzen binden. Die Primerschmelztemperatur für PCR-Versuche sollte in der Regel zwischen 55°C und 65°C liegen. Die Primerlänge sollte daher eine Länge von 30 Nukleotiden nicht übersteigen. Die Schmelztemperatur eines Primers hängt allerdings nicht nur von seiner Länge ab, sondern auch von seiner Nukleotidzusammensetzung. G-C-Basenpaarungen werden durch drei Wasserstoffbrücken stabilisiert, T-A-Basenpaarungen nur über zwei. G-C-Basenpaarungen sind also „stabiler“. Die Schmelztemperatur nimmt also mit der Anzahl an G-Nukleotiden und C-Nukleotiden zu. Die Schmelztemperatur lässt sich mit mehreren Methoden berechnen:
- die Wallace-Regel:
TM = (AT * 2) + (GC * 4) − 5
- die GC-Methode ist die einfachste aber auch ungenaueste Methode:
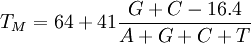
- die "salt adjusted"-Methode ist etwas genauer und bezieht die Konzentration an Na+-Ionen im Reaktionsansatz mit ein:
![T_M=100,5+41*\frac{C+G}{A+C+G+T}-\frac{820}{A+C+G+T}*16,6*\log_{10}([\mbox{Na}^+])](/pictures/dewiki/49/101ec424d16acca39d6aff08c9f1d12a.png)
- die komplizierteste Methode ist die base stacking Methode, bei der die Enthalpie- und terme der Helixbildung bei der Hybridisierung mit einbezogen werden:
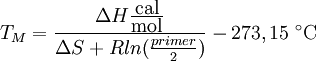
Mittlerweile gibt es aber eine große Anzahl an Software, mit der man die Schmelztemperatur von Primern berechnen kann.
Siehe auch
Weblinks
Wikimedia Foundation.
