- Tyrosinkinase ABL1
-
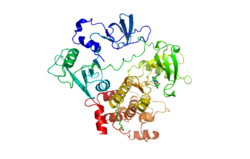
c-Abl Teilstruktur von c-Abl mit den Domänen SH2 (türkis), SH3 (blau) und der Kinasedomäne (grün-rot) in Komplex mit den Inhibitoren PD166326 und Myristylat basierend auf der Kristallstruktur PDB 2FO0 Masse/Länge Primärstruktur 1130 Aminosäuren Kofaktor Mg2+ oder Mn2+> Isoformen 1A, 1B Bezeichner Gen-Name(n) ABL1; c-Abl; ABL; JTK7; E430008G22Rik Externe IDs OMIM: 189980 UniProt: P00519 MGI: 87859 Enzymklassifikation EC, Kategorie 2.7.10.2 Tyrosinkinase Reaktionsart Phosphorylierung Substrat CRK, CRKL, DOK1 Vorkommen Übergeordnetes Taxon Landwirbeltiere Die Tyrosinkinase ABL1 (Akronym für Abelson murine leukemia viral oncogene homolog 1, auch c-Abl, p150) ist ein in der Zellmembran verschiedener Körperzellen vorkommendes Protein aus der Familie der Tyrosinkinasen.
Dieses Protein ist in vielen zellulären Prozessen, wie beispielsweise der Zellmigration, Zelladhäsion, Zelldifferenzierung sowie Apoptose involviert und ist ein wichtiges Element für die Signaltransduktion über den T-Zell-Rezeptor. c-Abl ist das Genprodukt des gleichnamigen Protoonkogens c-abl, einer Vorstufe eines potenziell krebsauslösenden Gens. Durch Austausch (Translokation) von Chromosomenfragmenten zwischen den c-abl beherbergenden Chromosom 9 und den bcr-Gen beherbergenden Chromosom 22, der zu dem sogenannten Philadelphia-Chromosom führen kann, entsteht ein neues bcr-abl-Gen, welches das zuerst gefundene durch Chromosomenveränderungen entstandene Onkogen war und unter Anderem bei 95% der chronischen myeloischen Leukämien (CML) nachgewiesen werden kann. Abl-Proteine sind daher beliebte Zielstrukturen für die Arzneistoffentwicklung.
Inhaltsverzeichnis
Biochemie
Struktur
c-Abl ist ein Protein mit einer Molekülmasse von etwa 145 kDa, das durch ein Gen auf dem Chromosom 9 Genlocus q34 codiert wird. Es besteht aus jeweils einer SH2- und SH3-Domäne, die für die Regulierung von c-Abl verantwortlich sind, und einer die Enzymfunktion tragenden Kinasedomäne. Der C-Terminus trägt Bindungsdomänen für eine Interaktion mit der DNA und mit Aktin. Durch unterschiedliches Splicing können zwei verschiedene N-terminale Proteinsequenzen gebildet werden. Der N-Terminus von c-Abl, der im Falle der Splicevariante 1B zusätzlich eine Myristylierungsstelle trägt, ist für die Autoinhibition verantwortlich. Eine Bindungsstelle für Myristinsäurereste konnte im C-Lobe der Kinasefunktion identifiziert werden[1].
 Vergleich der strukturellen Organisation von c-Abl (Splicevarianten 1a und 1b) mit BCR/Abl. (BD: Bindungsdomäne, NLS: Kernlokalisierungssignal, NES: Kernexportsignal)
Vergleich der strukturellen Organisation von c-Abl (Splicevarianten 1a und 1b) mit BCR/Abl. (BD: Bindungsdomäne, NLS: Kernlokalisierungssignal, NES: Kernexportsignal)
Aktivierung und Regulierung
c-Abl liegt in einem basal inaktiven Zustand vor. Für die Inaktivität wird der N-terminale Teil des Proteins und der Myristylrest verantwortlich gemacht, wenngleich die Spleißvariante 1A keine solche Myristylierungsstelle besitzt. An der Stabilisierung des inaktiven Zustands können jedoch auch weitere mit Fettsäuren modifizierte Proteine, wie beispielsweise Fus1, beteiligt sein [2]. Ein komplettes Fehlen des Terminus, wie beispielsweise im Falle des onkogenen viralen Abkömmlings v-Abl und der onkogenen Mutante BCR-Abl, wird mit einer konstitutiven Aktivität der Kinasefunktion und einem krebsauslösenden Potenzial in Verbindung gebracht.
c-Abl kann durch Rezeptor-Tyrosikinasen, wie beispielsweise der EGF-Rezeptor, und durch Nicht-Rezeptortyrosinkinasen, wie c-Src aktiviert werden [3].
Weiterführende Literatur
- Lin J., Arlinghaus R. (2008). Activated c-Abl tyrosine kinase in malignant solid tumors. Oncogene 27:4385-4391.
Einzelnachweise
- ↑ Nagar B. et al. (2003). Structural basis for the autoinhibition of c-Abl tyrosine kinase. Cell 112:859-871.
- ↑ Lin J. et al. (2007). Oncogenic activation of c-Abl in non-small cell lung cancer cells lacking FUS1 expression: inhibition of c-Abl by the tumor suppressor gene product Fus1. Oncogene 26:6989-6996.
- ↑ Srinivasan D., Plattner R. (2006). Activation of Abl tyrosine kinases promotes invasion of aggressive breast cancer cells. Cancer Res. 66:5648–5655.
Wikimedia Foundation.